Репродуктивно-Респираторный синдром свиней (PRRS) по-прежнему является, безусловно, болезнью связанной с самым большим экономическим ущербом в свиноводстве, и уровень его контроля далек от удовлетворительного. Лучшее и более полное представление о разновидностях PRRSV и мониторинг циркуляции новых штаммов в определенной области/стране/континенте, безусловно, поможет ветеринарам и производителям реализовать программы контроля и, возможно, эрадикации (искоренения). Для этого секвенирование PRRSV стало доступно по всему миру, начиная с конца 1990 'годов в основном в Северной Америке, Европе и Юго-Восточной Азии.
Геном PRRSV (см. рисунок 1) представляет одноцепочечную молекулу РНК, что делает его склонным "делать ошибки" (в виде генетической мутации) во время репликации в хозяине. Эта "тенденция к ошибкам" является причиной наличия различных prrsv штаммов, каждый из которых уникален в своей собственной генетической последовательности. Отвечают ли различия последовательности за разное (клинико-патологическое? иммунологическое?) поведение " - по-прежнему является предметом обсуждения среди практиков и исследователей.

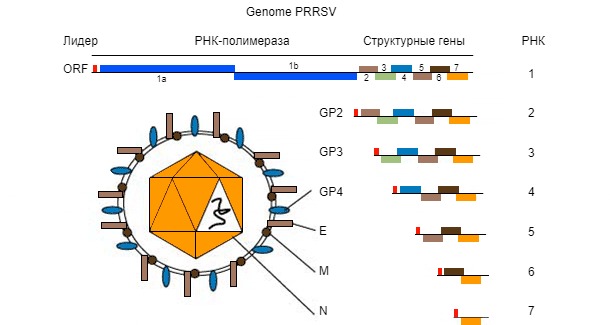
Рис. 1. Геном вируса РРСС - одноцепочная молекула РНА.
Основы секвенирования PRRS
Вирусное секвенирование производится на материалах ПЦР из полевых образцов (сыворотки, ткани, оральные жидкости). Чтение нуклеотидов осуществляется обычно из некоторых фрагментов генома вирусной РНК (см. рисунок 2) в целевых регионах – ORFs (открытая рамка считывания), а затем сравнивающих процент гомологии путем филогенетических анализов, выполняемых применением соответствующего программного обеспечения. Результат этого процесса возвращает степень сходства (гомологии) между различными штаммами PRRSV. С помощью графической визуализации программного обеспечения, а также дендрограммы (или" филогенетическое дерево"), определяют родство (или отсутствие родства) со ссылкой на вирусную последовательность (см. рисунок 3).

Рис. 2. Вирусное секвенирование производится на ПЦР-продуктах, путем чтения нуклеотидов из некоторых фрагментов генома вирусной РНК в целевых регионах-ORF.
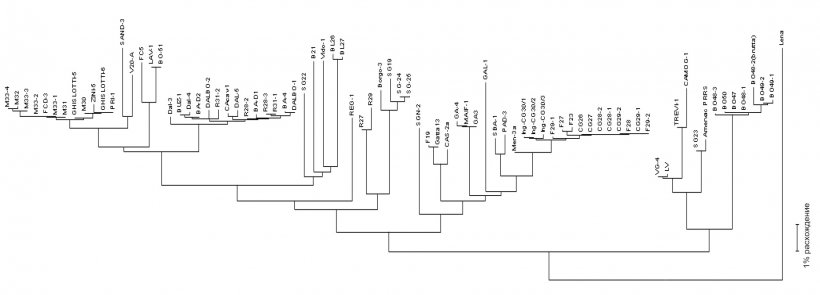
Рис. 3. Дендрограммы или филогенетическое дерево используются для графического представления степени сходства (гомологии) между различными штаммами к эталонной вирусной последовательности.
Геном PRRSV кодирует не менее дестяти ORF. Наиболее часто используемый для секвенирования – хотя они составляют только 4% и 3% всего генома соответственно - ORF5 (Кодировка негликозилированного белка Е) и ORF7 (кодирующий нуклеокапсид (N) белка). ORF5 представляет более переменную область, в то время как ORF7 представляет собой более постоянную область. Из-за этого та же степень вариации (т. е. 5% вариации), найденная в ORF7, более "драматична" – в плане генетической изменчивости-по сравнению с ORF5. Толкование сходства (т. е. вирусы связаны или нет) требует гораздо больше дополнительной информации, так как скорость генетических изменений может быть весьма вариабельной.
Чрезвычайно важно вести запись всех последовательностей, однозначно идентифицированных и аннотированных и тщательно зафиксировать дату, тип предприятия (ферма 1-2-3), движение животных, местоположение фермы (широта/Долгота GPS) и происхождение последовательности (Тип животного/ткань/образец). На сегодняшний день наш массив данных последовательностей PRRSV содержит более 1300 последовательностей ORF7, начиная с 2002 года. Для интерпретации и понимания различий еще более важно сопоставлять отдельные последовательности с клиническими случаями, характеризующимися количеством абортов у свиноматок и смертностью поросят перед отъёмом в типе 1 и уровень смертности в типах 2 и 3.
Практические вопросы
Часто задаваемые вопросы от ветеринаров и производственников:
- Представляют ли наблюдаемые генетические различия между последовательностями нормальную вариацию одноного штамма PRRSV в ферме / системе или они представляют собой несколько различных штаммов, присутствующих на ферме одновременно или за короткий промежуток времени?
- У меня сейчас" новая вспышка, вызвана новым штаммом или это рециркуляция?
Чтобы ответить на эти вопросы, мы должны договориться о принятой степени гомологии между двумя вирусными штаммами, собранными в течение определенного периода времени (12-24 месяцев?). Иными словами, сходство считается установленным при 97-98% гомологии последовательности или 2-3% разница. Это является общепринятым значением. Согласно моему опыту, довольно трудно увидеть изменение выше 2% в клинически стабильной закрытой популяции "(традиционный тип фермы с соответствующим движением животных), поскольку мы наблюдали получение" того же штамма " в течение периода до 3 лет в одном и том же клинически стабильном стаде. Напротив этого, каждый раз, когда мы замечали консистентную активность репродуктивно-респираторного синдрома свиней "нового" и филогенетически различных (90% гомологии или меньше) штамм восстанавливается. К сожалению, мы точно не знаем, являются ли эти большие различия, которые мы иногда наблюдаем, результатом внезапного изменения вируса/мутации (по моему личному мнению маловероятно) или внедрения нового штамма. Хорошо признан тот факт, что генетическое сходство / разнообразие никоим образом не предсказывает иммунологическое сходство (т. е. существование перекрестного иммунитета) и не позволяет предсказать внутреннеприсущей патогенности (не говорит о вирулентности определенного штамма).
Полностью расшифрованные последовательности генома, которые сегодня доступны (к сожалению, больше для научных целей, чем для повседневного использования в диагностике), безусловно, помогут ответить на этот вопрос.
Очень важно проанализировать новые последовательности PRRSV в сопоставлении с широким справочным набором, представляющим ферму, систему и регион, а также последовательности имеющихся коммерческих вакцин (это позволит провести различие между полевыми и вакцинными штаммами). На данный момент мы все еще используем набор программного обеспечения с открытым исходным кодом, управляемый университетом Падуи (Padova University), чтобы построить наши филогенетические деревья и сохранить их организованным в зависимости от движения свиней в нашей общей производственной системе. Мы могли бы также присоединится к двум другим "специальных компьютерных программ" (Биопортал университета Дэвиса Калифорнии и CLASSIFARM- PATH от IZSLER (Брешиа, Италия), что будет иметь гораздо больший набор последовательностей для сравнения. Это позволяет нам лучше понять PRRSV распространение в Италии и, возможно, в Евросоюзе.

Благодарность: Спасибо профессору Микеле Дриго (UNI-PD) за интересное обсуждение и рецензию этой публикации.



